甘草黄酮合酶Ⅱ催化甘草素特异性合成7,4′-二羟基黄酮
黄酮类化合物是植物次级代谢产物的主要种类之一,包括9000多种结构[1],分别属于黄酮、黄烷酮、异黄酮、查尔酮、黄酮醇、二氢黄酮醇、黄烷醇、花青素以及他们的衍生物。其中黄酮是指具有2-苯基苯并γ吡喃酮分子结构且在2号和3号碳原子之间具有碳碳双键的一类化合物(图1),因具有抗氧化[2]、抗炎、抗癌[3-5]、防治心脑血管疾病[6]、预防肥胖和糖尿病[1]、抗凝血[7]等众多药理作用而受到人们的广泛关注。例如,7,4′-二羟基黄酮(DHF)(图1)是一种存在于甘草中的黄酮化合物[8],具有活血化瘀、镇痛抗炎[9]、抗氧化[10]、抗真菌[11]、抑制嗜酸性粒细胞趋化因子产生[11]、抑制MUC5A基因表达和黏液产生[10]等生理和药理活性,在临床治疗和医疗保健领域具有巨大的应用价值。
图1
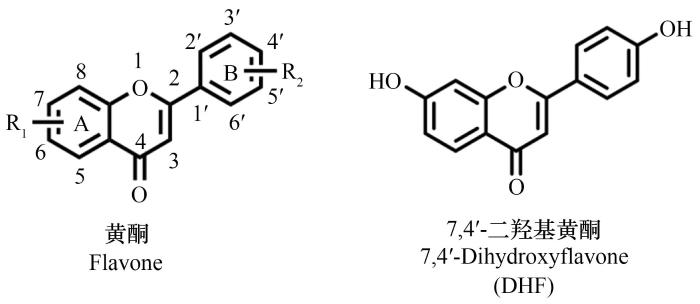
图1 黄酮的结构
Fig.1 The structure of flavone
当前制备黄酮类化合物的方法主要有植物提取法[12]、化学合成法[13]以及生物合成法[14-15]等,尤其是近年来利用微生物细胞工厂合成了诸多植物天然产物[16-18]。其中,在合成7,4′-二羟基黄酮的方法中以生物催化为代表的生物法因其具有操作简便、成本低、反应条件温和等优点[19-20]备受青睐。7,4′-二羟基黄酮(DHF)的生物催化合成中主要涉及两种类型的酶,黄酮合酶(FNS) Ⅰ和Ⅱ可催化甘草素合成7,4′-二羟基黄酮(图2)[21]。其中,FNS Ⅰ是一种依赖于α-酮戊二酸的双加氧酶,主要存在于欧芹、圆叶当归、小茴香等伞形科植物中[22-23];FNS Ⅱ是一种依赖于烟酰胺腺嘌呤二核苷酸磷酸(NADPH)的单加氧酶(表1,图2),广泛存在于大豆、苜蓿、丹参等豆科植物中[31]。例如,大豆来源的FNS Ⅱ(CYP93B16)被挖掘且被证明可以催化甘草素、柚皮素、圣草酚合成相应的黄酮化合物(图2)[21]。然而,富含7,4′-二羟基黄酮的豆科植物甘草[8]的FNS Ⅱ仍未见报道。因此,本研究拟基于甘草的转录组学分析、氨基酸序列比对以及分子进化树分析从甘草中挖掘新的FNS Ⅱ并表征其催化功能;利用蛋白质结构预测、分子对接和分子动学模拟探究其催化机制;最后,采用条件优化和过程强化的方式构建其最佳细胞催化工艺。
图2
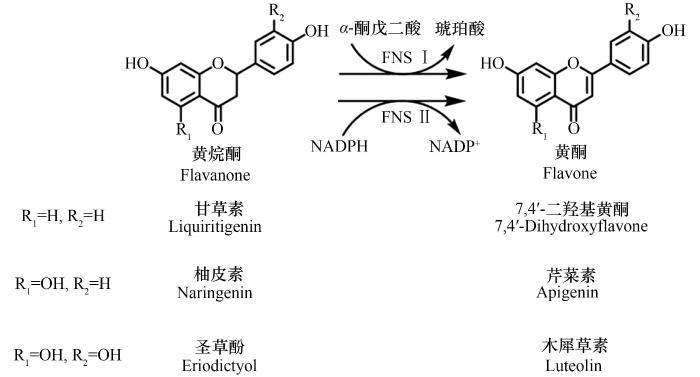
图2 黄酮合酶催化黄烷酮合成黄酮
Fig.2 Flavonoid synthase catalyzes flavanone to synthesize flavone
表1 不同FNS Ⅰ和FNS Ⅱ的对比
Table 1
| 名称 | 植物来源 | 类型 | 米氏常数(底物) | 文献 |
|---|---|---|---|---|
| CYP93G1 | 水稻 | FNS Ⅱ | 3.2 µmol/L (柚皮素) | [24] |
| 1.5 µmol/L (圣草酚) | ||||
| CYP93B6 | 紫苏 | FNS Ⅱ | 8.8 µmol/L (柚皮素) | [25] |
| 11.9 µmol/L (圣草酚) | ||||
| CYP93B16 | 大豆 | FNS Ⅱ | 4.2 µmol/L (甘草素) | [21] |
| 2.5 µmol/L (柚皮素) | ||||
| 1.8 µmol/L (圣草酚) | ||||
| CYP93B24 | 黄芩 | FNS Ⅱ | 0.24 µmol/L (松属素) | [26] |
| 0.28 µmol/L (柚皮素) | ||||
| CYP93B25 | 黄芩 | FNS Ⅱ | 0.46 µmol/L (松属素) | [26] |
| LjFNSⅡ-1.1 | 金银花 | FNS Ⅱ | 6.48 µmol/L (甘草素) | [27] |
| 9.93 µmol/L (柚皮素) | ||||
| 5.07 µmol/L (圣草酚) | ||||
| LjFNSⅡ-2.1 | 金银花 | FNS Ⅱ | 2.56 µmol/L (甘草素) | [27] |
| 1.63 µmol/L (柚皮素) | ||||
| 2.05 µmol/L (圣草酚) | ||||
| LmFNSⅡ-1.1 | 灰毡毛忍冬 | FNS Ⅱ | 2.38 µmol/L (甘草素) | [27] |
| 1.63 µmol/L (柚皮素) | ||||
| 3.09 µmol/L (圣草酚) | ||||
| PaFNSI | 鼠尾草 | FNS Ⅰ | 19.91µmol/L (柚皮素) | [28] |
| PcFNSI | 欧芹 | FNS Ⅰ | 5.00 µmol/L (柚皮素) | [29] |
| 8.00 µmol/L (圣草酚) | ||||
| OsFNSI | 水稻 | FNS Ⅰ | 20.00 µmol/L (柚皮素) | [30] |
新窗口打开| 下载CSV
1 材料与方法
1.1 材料
(1)菌株:克隆宿主大肠杆菌(Escherichia coli DH5α)和克隆载体(pUC19)以及表达宿主酵母细胞Saccharomyces cerevisiae BY4742均为实验室保藏。
(2)酶制剂:限制性内切酶(BamHI, SalI)购买自NEB公司;高保真Phanta Max Super-Fidelity DNA聚合酶等均购买自南京诺唯赞生物科技股份有限公司。
(3)生化学试剂:异丙基-β-D-硫代半乳糖苷(IPTG);氨苄青霉素、遗传霉素、鲑鱼精DNA均购买自索莱宝生物科技有限公司;聚乙二醇3350 (PEG3350)、无水醋酸锂、甘草素,7,4′-二羟基黄酮、氨基酸粉末、三羟甲基氨基甲烷(Tris)等均购买自北京伊诺凯科技有限公司;酵母提取物、胰蛋白胨购买自英国OXOID公司。
(4)试剂盒:胶回收试剂盒购买自赛默飞世尔科技(中国)有限公司;质粒提取试剂盒等购买自天根生化科技(北京)有限公司;其他试剂购买自国药集团化学试剂有限公司。
(5)所构建的酿酒酵母菌株及基因型如表2所示。
表 2 酵母菌株及其基因型
Table 2
| 菌株名称 | 基因型 |
|---|---|
| Yeast | BY4742,MATα his3Δ1 leu2Δ0 lys2Δ0 ura3Δ0 |
| Yeast-F01 | BY4742,YPRCtau3::PTDH3-Gur.gene26505-TPGK1-leu2 |
| Yeast-F02 | BY4742,YPRCtau3::PTDH3-Gur.gene26116-TPGK1-leu2 |
| Yeast-F03 | BY4742,rDNA::PTDH3-Gur.gene26505-TPGK1- KanMX |
| Yeast-F04 | Yeast-F04,YPRCtau3::PTDH3-Gur.gene26505-TPGK1- leu2 |
新窗口打开| 下载CSV
1.2 菌株培养
(1)大肠杆菌培养:挑取重组大肠杆菌单菌落,接种到含有适宜抗生素(100 mg/L氨苄青霉素 )的固体培养基上;过夜培养后挑取菌落进行菌落PCR验证;验证成功后挑取菌落接种到含有适宜抗生素的LB液体培养基培养,提取质粒测序验证和扩增目的基因表达盒。
(2)酿酒酵母培养:挑取重组酿酒酵母单菌落,接种到含有适宜抗生素和营养缺陷型的固体培养基中;在培养箱中培养2~3 d挑取菌落裂解后进行菌落PCR验证;验证成功后挑取正确单菌落接种到含有适宜抗生素和营养缺陷型的液体培养基中,培养24 h,按照10%比例接种到YPD液体培养基中,培养相应时间,添加底物进行催化反应。
1.3 细胞催化反应与活性检测
将表达目的蛋白的酿酒酵母接种至3 ml YPD液体培养基中30℃、250 r/min培养24 h得到种子液,按照10%的接种比例接种至30 ml YPD液体培养基中培养12 h加入终浓度 为1 mmol/L的底物,在30℃、250 r/min的条件下反应48 h后,萃取产物,测定细胞干重和产物浓度。细胞催化活性以1 g/L的细胞干重(CDW)合成的7,4′-二羟基黄酮的浓度为衡量指标,细胞催化效率以底物甘草素转化率为衡量指标。
1.4 蛋白质结构预测与分子动力学模拟
利用AlphaFold2[32]进行蛋白质结构预测,AutoDock Vina[33]进行分子对接(配体结构下载于PubChem网站);利用CHARMM-GUI网站(
1.5 催化工艺优化与强化
(1)目的基因过表达:使用不同酿酒酵母拷贝数位点(单拷贝位点YPRCtau3,多拷贝位点rDNA以及两个位点同时整合)进行外源基因整合构建表2所示的工程菌,分别在酵母培养时间为12 h时添加终浓度为1 mmol/L的甘草素底物,在30℃、250 r/min的条件下反应48 h,考察目的基因Gur.gene26505过表达对甘草素转化率的影响。
(2)底物添加时间优化:分别在酵母接种后的0~84 h时添加终浓度为1 mmol/L的甘草素底物,在30℃、250 r/min的条件下反应48 h,考察不同底物添加时间对甘草素转化率的影响。
(3) 催化时间优化:将酵母培养最优时间,添加终浓度为1 mmol/L的甘草素底物,在30℃、250 r/min的条件下反应12~96 h,考察不同催化时间对甘草素转化率的影响,确定最佳催化时间。
(4)过程强化:在以上最佳工艺下,每隔12 h分别补加终浓度分别为初始培养基浓度的酵母粉、蛋白胨、葡萄糖,考察培养基成分强化对催化过程中甘草素转化率的影响。
1.6 HPLC和LC-MS分析检测
取1 ml反应液加入2倍体积乙酸乙酯混匀萃取,吸取1 ml萃取液,蒸干,加入1 ml甲醇重新溶解,用0.22 μm的有机滤头过滤到进样瓶中,采用Agilent 1260高效液相色谱测定浓度。检测条件:色谱柱为Agilent Poroshell 120 EC-C18 (2.7 μm,3.0×100 mm);流动相A为0.1%(体积)的甲酸水溶液;流动相B为乙腈;梯度洗脱,1~20 min:(20% B~100% B(体积)),20~25 min:(100% B(体积)),25~26 min:(100% B~20% B(体积)),26~30 min:(20% B(体积));流速为0.5 ml/min;柱温35℃;紫外吸收检测器波长为325 nm。
质谱检测采用岛津液相色谱-四极杆质谱联用仪(LCMS-8040),液相条件与HPLC相同,质谱条件:雾化气流速N23.0 L/min,电喷雾离子源(ESI),加热块温度400℃,Q3正离子扫描模式,m/z测定范围100~400,测定时间0~30 min,扫描速度909 u/s。
2 结果与分析
2.1 基于转录组分析的甘草黄酮合酶挖掘
由于胀果甘草的基因组尚未解析,为了获得甘草的FNS Ⅱ建立了胀果甘草的转录组分析方案。以已报道的大豆来源的FNS Ⅱ(CYP93B16)[21]为模板对胀果甘草转录组进行氨基酸序列同源比对分析共获得214条候选序列。序列同源比对分析发现只有四条基因:Gur.gene26505、Gur.gene26116、Gur.gene26926、Gur.gene26365与CYP93B16[21]的氨基酸序列相似度在45%以上(表3),且将214条候选序列与已知功能的甘草[34]、大丁草[35]、苜蓿[36]等来源的CYP93B家族的细胞色素P450(表4)构建分子进化树(图3)表明这四条基因均属于CYP93B家族的细胞色素P450。因此,Gur.gene26505、Gur.gene26116、Gur.gene26926和Gur.gene26365被确定为具有潜在功能的黄酮合酶。同时,分子进化树也表明Gur.gene26505属于黄酮合酶Ⅱ分支可催化黄烷酮只生成黄酮[21],而Gur.gene26116、Gur.gene26926和Gur.gene26365属于黄酮2位羟化酶(F2H)分支催化黄烷酮生成黄酮和2-羟基黄烷酮[34](图4)。
表3 候选基因信息
Table 3
| 基因名称 | 核酸序列长度/bp | 与CYP93B16 氨基酸序列相似度/% |
|---|---|---|
| Gur.gene26505 | 1572 | 77 |
| Gur.gene26116 | 1593 | 51 |
| Gur.gene26926 | 1221 | 47 |
| Gur.gene26365 | 1572 | 45 |
新窗口打开| 下载CSV
表4 不同来源CYP93B家族的基因
Table 4
| 名称 | 植物来源 | GenBank | 类型 | 文献 |
|---|---|---|---|---|
| CYP93B1 | 刺果甘草 | P93149 | 黄酮2位羟化酶 | [34] |
| CYP93B2 | 大丁草 | AAD39549 | 黄酮合酶Ⅱ | [35] |
| CYP93B3 | 金鱼草 | BAA84071 | 黄酮合酶Ⅱ | [34] |
| CYP93B4 | 矮牵牛 | BAA84072 | 黄酮合酶Ⅱ | [34] |
| CYP93B5 | 翠菊 | AAF04115 | 黄酮合酶Ⅱ | [21] |
| CYP93B6 | 紫苏 | BAB59004 | 黄酮合酶Ⅱ | [26] |
| CYP93B11 | 苜蓿 | ABC86159 | 黄酮2位羟化酶 | [36] |
| CYP93B12 | 苜蓿 | DQ335809 | 黄酮合酶Ⅱ | [36] |
| CYP93B13 | 三花龙胆 | BAD91809 | 黄酮合酶Ⅱ | [37] |
| CYP93B14 | 山柳菊 | ACB56919 | 黄酮合酶Ⅱ | [38] |
| CYP93B16 | 大豆 | FJ767774 | 黄酮合酶Ⅱ | [21] |
| CYP93B17 | 六倍利 | BAF49323 | 黄酮合酶Ⅱ | [39] |
| CYP93B18 | 茶树 | ACH99109 | 黄酮合酶Ⅱ | [21] |
新窗口打开| 下载CSV
图3
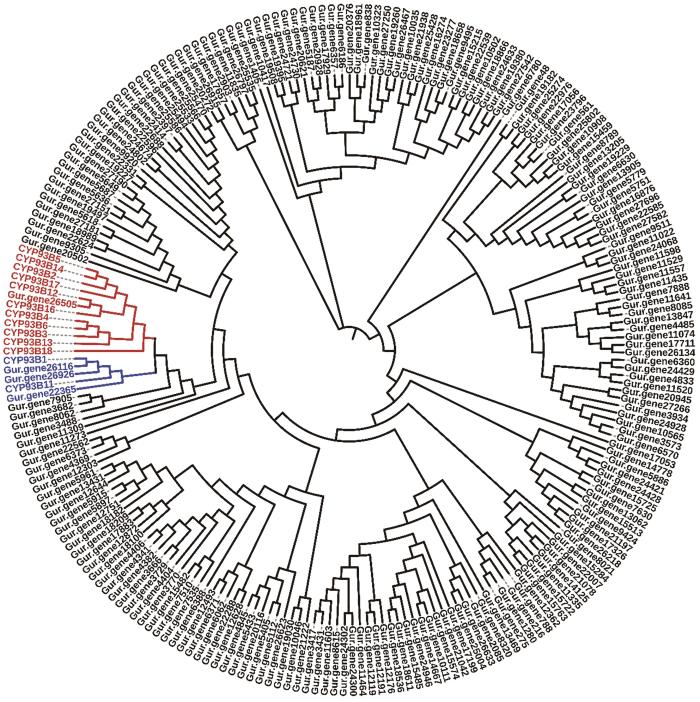
图3 进化树分析
Fig.3 Evolutionary tree analysis
图4

图4 黄酮合酶Ⅱ和黄酮2位羟化酶功能比较
Fig.4 Comparison of flavonoid synthase Ⅱ and flavonoid 2-hydroxylase
2.2 候选黄酮合酶的催化功能表征
为了表征4个候选基因的功能,通过对甘草根的总RNA的提取、反转录构建其cDNA文库。以cDNA文库为模板,成功扩增了Gur.gene26505和Gur.gene26116基因并测序验证序列正确。构建其酿酒酵母细胞表达系统,分别命名为Yeast-F01和Yeast-F02。通过外源添加甘草素的方式进行细胞催化反应,检测结果如图5所示,结果表明重组菌Yeast-F01在外源添加了底物甘草素之后,以甘草素为底物特异性生成了7,4′-二羟基黄酮,而Yeast-F02的产物峰除7,4′-二羟基黄酮之外还有两个产物峰(2-羟基甘草素及其互变异构体)[34](图5)。表明Gur.gene26505可催化甘草素特异性合成7,4′-二羟基黄酮,属于新的甘草黄酮合酶Ⅱ,而Gur.gene26116的产物较多、特异性较差,属于黄酮2位羟化酶。
图5
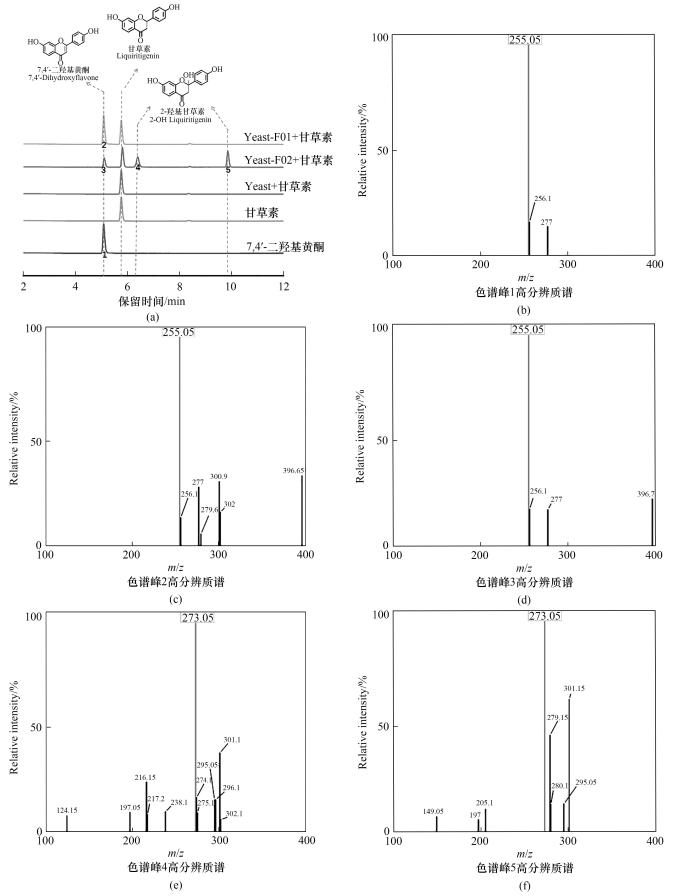
图5 甘草黄酮合酶Ⅱ候选基因的功能表征
Fig.5 Functional characterization of candidate genes of flavone synthase Ⅱ
为了表征2个工程菌(Yeast-F01和Yeast-F02)合成7,4′-二羟基黄酮的能力,分别添加相同浓度(1 mmol/L)的甘草素反应48 h测定7,4′-二羟基黄酮的产量。以1 g的干细胞合成的7,4′-二羟基黄酮质量(mg)为衡量指标对2个工程菌株的合成能力进行了评估。结果如图6所示,表达Gur.gene26505的工程菌Yeast-F01底物转化率达17.44%,同时其细胞催化活性达3.4 mg/g,是Yeast-F02细胞催化活性的2倍。因此,表达Gur.gene26505的工程菌Yeast-F01除了具有产物特异性的优点之外其催化活性也高,但是其底物转化率相对较低。
图6
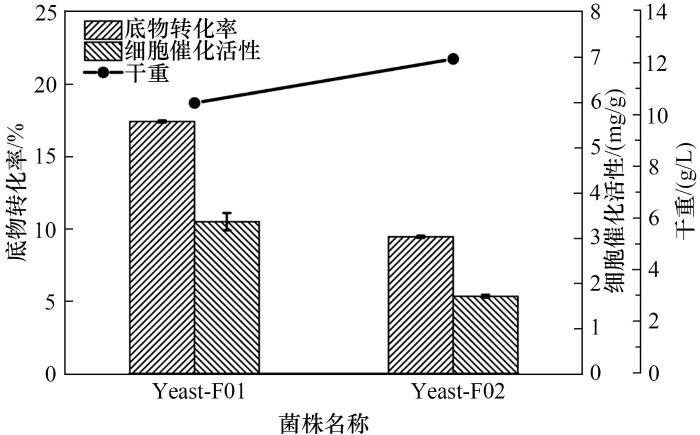
图6 工程菌合成7,4′-二羟基黄酮能力评估
(细胞催化活性(mg/g)=7,4′-二羟基黄酮的浓度(mg/L)/细胞干重(g/L);甘草素转化率(%)=7,4′-二羟基黄酮的浓度(mmol/L)/甘草素初始添加量(mmol/L))
Fig.6 Evaluation of the ability of engineering bacteria to synthesize 7,4'-dihydroxyflavone
2.3 黄酮合酶Ⅱ(Gur.gene26505)特异性催化机理探究
为了探究Gur.gene26505和Gur.gene26116催化甘草素产物差异的原因,利用AlphaFold2对Gur.gene26505和Gur.26116的蛋白质结构进行了预测,并将中间羟化产物2-羟基甘草素与其进行分子对接和100 ns的分子动力学模拟(图7),发现Gur.gene26505的蛋白结构中312位天冬氨酸的氧原子与2-羟基甘草素3号位的氢原子形成了氢键相互作用可将其夺走,并且315位苏氨酸的氢原子与2-羟基甘草素2号位的羟基氧原子形成了氢键相互作用[图8(a)],使其容易离去从而生成碳碳双键得到最终产物7,4′-二羟基黄酮;Gur.gene26116结构中同样具有300位天冬氨酸和303位苏氨酸的脱水活性中心,使其具有合成7,4′-二羟基黄酮的能力,但是其117位的大位阻苯丙氨酸残基处在一个柔性loop结构上[图8(d)],由于柔性loop往里收缩导致117位的苯丙氨酸靠近羟化中心阻碍了羟化产物2-羟基甘草素的位置转变[图8(b)],从而使部分羟化产物2-羟基甘草素无法进入脱水中心导致Gur.gene26116产物复杂。相反,Gur.gene26505活性口袋附近特有的刚性结构β片层[图8(c)]使大位阻苯丙氨酸残基翻转至羟化中心下方,消除了羟化产物2-羟基甘草素进入脱水中心的阻力,进而发生C2-C3位的脱水反应特异性生成7,4′-二羟基黄酮[图8(a)]。
图7

图7 Gur.gene26505和Gur.gene26116分子动力学模拟的均方根偏差数据
Fig.7 The root mean square deviation data for molecular dynamics simulations of Gur.gene26505 and Gur.gene26116
图8
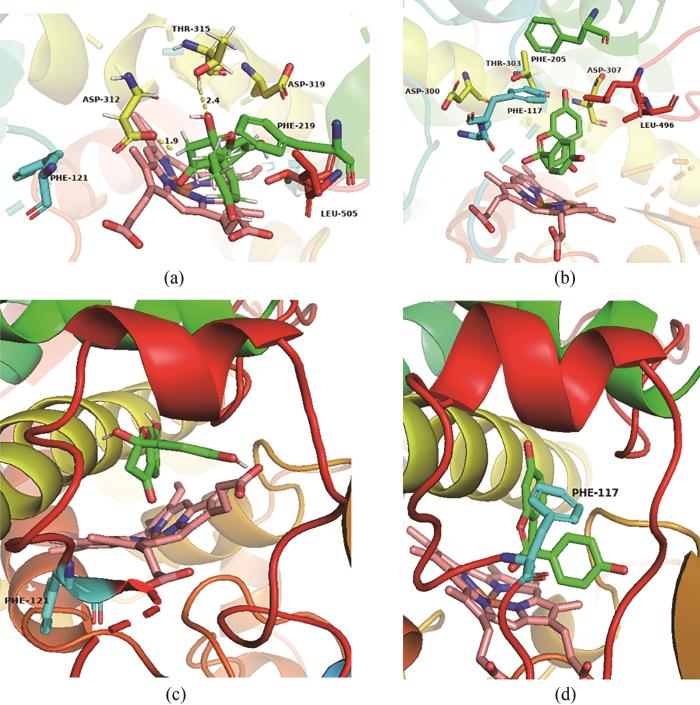
图8 Gur.gene26505特异性催化机理
Fig.8 Specific catalytic mechanism of Gur. gene26505
2.4 高效合成7,4′-二羟基黄酮的细胞催化工艺
为了实现7,4′-二羟基黄酮的高效合成,采取了目的基因Gur.gene26505过表达,优化甘草素添加时间、催化时间以及强化细胞生长过程的策略。首先选择可催化甘草素特异性合成7,4′-二羟基黄酮的Gur.gene26505分别构建了其在酿酒酵母的单拷贝、多拷贝位以及单拷贝和多拷贝位点同时整合的重组菌Yeast-F01、Yeast-F03和Yeast-F04,外源添加相同浓度(1 mmol/L)的甘草素反应48 h测定甘草素的转化率。结果如图9(a)所示,随着Gur.gene26505整合位点拷贝数的增加,底物转化率逐渐升高,Gur.gene26505过表达的重组菌株Yeast-F04转化率上升至29.40 %,是重组菌Yeast-F03和Yeast-F01的1.5倍和1.7倍,表明过表达Gur.gene26505可以一定程度提高底物转化率。
图9

图9 条件优化提高底物转化率
Fig.9 Conditional optimization to improve substrate conversion
其次,考察了不同底物添加时间、催化时间对底物转化率的影响。结果表明随着底物添加时间点的延后,底物转化率呈现下降趋势,在0~36 h 之内添加底物时底物转化率维持在25%以上,其中在6 h时底物转化率最高达到33.57%[图9(b)]。另外发现随着催化反应时间的延长,底物甘草素的转化率呈现上升趋势,最高达50.20%[图9(c)],但是后期84~96 h几乎趋于平稳并且细胞干重在60 h之后不再增加,细胞的生长状态和生物量限制了底物转化率的进一步上升。
因此,基于酵母细胞生长状态对底物转化率的影响,为了使得整个催化过程酵母全细胞具有高的生物量进一步提高底物转化率,对培养基成分酵母粉、蛋白胨、葡萄糖进行过程添加强化菌体生长,从接种开始每隔12 h分别添加培养基成分至其初始浓度,结果[图9(d)]表明酵母粉的过程添加会促进酵母细胞的生长,同时使底物的转化率提高,最高转化率在84 h可以达到76.67%,并且缩短了催化的时间,提高了催化工艺的效率。
进一步分析发现,0~36 h内添加底物以及通过强化菌体生长都可提高底物转化率的原因在于,这些策略可以使细胞具有良好的生长状态,从而为Gur.gene26505催化的反应(图2)提供充足的辅因子NADPH的供应,从而提高底物转化率。同时,过表达Gur.gene26505和延长反应时间也为催化过程提供了充足的催化剂和底物识别反应的时间。因此最佳催化工艺的建立和酶的催化特性息息相关。
综上所述,结合酶的催化特性选用过表达Gur.gene26505的工程菌Yeast-F04在接种后6 h添加1 mmol/L的甘草素作为底物,反应84 h,期间每隔12 h补加终浓度为1%的酵母粉,最终底物转化率可达到76.67%。
3 结论
(1)利用氨基酸序列同源比对以及分子进化树分析成功从胀果甘草中挖掘并克隆到2个黄酮合酶:Gur.gene26505和Gur.gene26116。
(2)利用细胞催化的方式成功实现了酿酒酵母细胞催化甘草素合成7,4′-二羟基黄酮。构建了Gur.gene26505和Gur.gene26116的酿酒酵母表达系统,结合分子进化树分析通过外源添加甘草素表征发现Gur.gene26505属于黄酮合酶Ⅱ,Gur.gene26116属于黄酮2位羟化酶。
(3)探究了Gur.gene26505和Gur.gene26116催化产物不同的原因。利用蛋白质结构预测、分子对接以及分子动力学模拟发现,Gur.gene26505催化产物单一的原因是活性口袋附近特有的刚性结构β片层使大位阻苯丙氨酸残基翻转至羟化中心下方,消除了羟化产物2-羟基甘草素进入脱水中心的阻力,进而发生C2-C3位的脱水反应特异性生成7,4′-二羟基黄酮。
(4)通过细胞催化体系条件优化和细胞生长强化,构建了高效合成7,4′-二羟基黄酮的细胞催化工艺。以单倍体酿酒酵母(BY4742)作为宿主菌,过表达甘草源黄酮合酶Ⅱ,在接种6 h后添加1 mmol/L的底物甘草素,并且通过强化菌体生长,反应84 h后,甘草素的转化率可达76.67%。本研究为7,4′-二羟基黄酮及其他黄酮类化合物的绿色生物制造提供了新的选择和理论指导。
- 我用了一个很复杂的图,帮你们解释下“23版最新北大核心目录有效期问题”。
- 重磅!CSSCI来源期刊(2023-2024版)最新期刊目录看点分析!全网首发!
- CSSCI官方早就公布了最新南核目录,有心的人已经拿到并且投入使用!附南核目录新增期刊!
- 北大核心期刊目录换届,我们应该熟知的10个知识点。
- 注意,最新期刊论文格式标准已发布,论文写作规则发生重大变化!文字版GB/T 7713.2—2022 学术论文编写规则
- 盘点那些评职称超管用的资源,1,3和5已经“绝种”了
- 职称话题| 为什么党校更认可省市级党报?是否有什么说据?还有哪些机构认可党报?
- 《农业经济》论文投稿解析,难度指数四颗星,附好发选题!
- 期刊知识:学位论文完成后是否可以拆分成期刊论文发表?
- 号外!出书的人注意啦:近期专著书号有空缺!!

 0373-5939925
0373-5939925 2851259250@qq.com
2851259250@qq.com



