基于近红外光谱结合化学计量学的转基因大豆产地判别
大豆起源于中国,营养丰富,可作为一种理想的食物,在世界上很多国家和地区的饮食中受到青睐,是人们不可获缺的食物。据中国海关总署统计2020年我国大豆进口总量超过1亿t,已成为最大的大豆进口国[1]。进口大豆大多为转基因大豆,其粗蛋白、脂肪和黄酮含量较高[2]。但是转基因大豆的潜在风险和危害是无法预测的,如对生物多样性的影响,对人类和动物健康的影响。因此我国对转基因大豆管控严格。目前我国的进口大豆来源于美洲国家,主要进口源有美国、巴西,阿根廷和加拿大。近年来我国从美国进口转基因大豆的数量急剧下降,调整为从美洲其他国家进口大豆。国外有部分商贩从美国进口转基因大豆,而后冒充本国大豆高价出口到我国,牟取不正当利益的同时给我国海关对转基因大豆检测和分类造成困难。国内有部分不法分子走私转基因大豆而后销售到国内市场,这对我国的生物安全带来了严重的威胁,因此对转基因大豆的产地朔源有利于从源头打击不法分子的犯罪行为,保护我国的生物安全。
转基因大豆检测和鉴别的主要方法是蛋白质检测方法与核酸检测方法[3]。蛋白质检测法主要包括试纸条法和酶联免疫吸附法[4-5];核酸检测方法主要包括定性PCR和环介导等温扩增技术[6-7]。以上检测方法是破坏性检测方法,需大量的实验试剂、试验过程繁琐复杂、投入的成本较高、检测专业性强、不易普及并且不能实现实时在线检测。
近红外光(near infrared,NIR)是介于可见光(visible,Vis)和中红外(mid infrared,MIR)之间的电磁辐射波。采用NIR光谱技术分析待测样品具有简单、高效、无损、实时、绿色环保的优点[8]。但是,NIR光谱受环境和样品影响较大,容易形成未知组分和灰色体系且有多重共线性问题[9]。化学计量学具有独特的优势,通过对样品测量数据的分析,可以最大限度的呈现出样品的各种化学信息。NIR光谱结合化学计量学是一种快速、准确、高效,可实现实时在线检测的方法[10]。
近年来,很多学者对NIR光谱结合化学计量学进行研究,NIR光谱结合化学计量学已广泛应用到食品、农业、医药、化工等多个领域[11-13]。其在鉴别转基因大豆和非转基因大豆上也成功应用,但在转基因大豆的产地判别上鲜有报道,针对我国进口转基因大豆的现状,对转基因大豆的朔源具有重要意义。
1 材料与方法
1.1 实验材料
试验选取转基因大豆分别为阿根廷转基因大豆MON89788品系(“A1”)、巴西转基因大豆MON89788品系(“B1”)、美国转基因大豆MON89788品系(“M1”)、加拿大转基因大豆MON89788品系(“J1”)以上4种大豆为同一品系不同产地的转基因大豆。“A1”,“M1”分别取50份样品,“B1”,“J1”分别取80份样品。如图1所示,从左到右分别为“A1”、“B1”、“M1”和“J1”单粒大豆特征,单粒大豆在外观上无明显差异,所有进口转基因大豆都由秦皇岛海关提供。
a-“A1”;b-“B1”;c-“M1”;d-“J1”
图1 四种转基因大豆单粒特征
Fig.1 Single grain characteristics of four transgenic soybeans
1.2 仪器与设备
全波反射型NIR光谱仪,检测器为Si和InGaAs,光谱扫描范围400~2 600 nm,北京伟创英图科技有限公司;ME204E电子天平,梅特勒-托利多有限公司;台式真空干燥箱DZF-6050,上海捷呈实验仪器有限公司。
1.3 实验方法
1.3.1 NIR光谱采集与光谱预处理
NIR光谱仪开机预热,白板校正后采集光谱。分别取4种转基因大豆共计260份样品,放置在干燥箱内,干燥温度为40 ℃,时间为36 h,光谱采集在(23±2)℃的恒温室内进行,试验所取的光谱范围为911~2 600 nm,每扫描10次计算1次平均光谱作为1条原始光谱。每间隔1 nm记录1个点,每条原始光谱记录了1 690个吸光度值。4种转基因大豆共采集了260条NIR光谱。
试验过程中由于实验仪器、环境和样品的影响,原始光谱中包含一部分的噪音。因此NIR光谱数据分析之前要对NIR原始光谱进行预处理,以此减少或者消除噪音对试验的影响。平滑是一种提高光谱信噪比的方法,原始光谱经过平滑处理后可有效减少光谱噪音。标准正态变量变换(standard normal variate transformation,SNV)主要是用来消除固体颗粒大小、表面散射以及光程变化对光谱的影响。本试验采取的光谱预处理方法为平滑+SNV[14]。
1.3.2 样品的划分和编号
Kennard-Stone(KS)算法,是在总样本中选出训练集样品,首先选择欧氏距离最远的2个样品进入训练集,其后通过计算剩下的每1个样品到训练集内每1个已知样品的欧式距离,找到拥有最大最小距离的待选样品放入训练集,以此类推,直到达到所要求的样品数目[15]。本试验共有转基因大豆样品260份,分别选取“A1”样品45份、“B1”样品75份、“M1”样品45份、“J1”样品75份共240份用来建立判别模型,剩余20份样品作为模型验证集。采用KS算法选择模型的训练集180份样品和预测集60份样品。分别对训练集、预测集和验证集样品编号,训练集中“A1”编号为X1~X34,“B1”编号为X35~X90,“M1”编号为X91~X124,“J1”编号为X125~X180。预测集中“A1”编号为Y1~Y11,“B1”编号为Y12~Y30,“M1”编号为Y31~Y41,“J1”编号为Y42~Y60。验证集中“A1”编号为Z1~Z5,“B1”编号为Z6~Z10,“M1”编号为Z11~Z15,“J1”编号为Z16~Z20。
1.3.3 主成分分析(principal component analysis,PCA)
PCA方法作为化学计量学中分析NIR光谱数据的常用方法,其核心思想是利用方差最大原则,对光谱数据多个自变量进行线性拟合。这样就可使高维的原始光谱数据最大限度的保留有效信息投影到低维空间,从而实现了光谱数据的降维,实现数据结构简化。在实际应用中取前面几个主成分,前面几个主成分基本包含了样品的绝大多数信息,可计算主成分的累计贡献率,当贡献率达到试验的要求时即可,这样可去除多余的数据,用更少的数据表达样品更多的信息,减少模型的计算量[16-17]。
1.3.4 偏最小二乘判别分析(partial least squares-discriminate analysis,PLS-DA)
PLS-DA本质上是一种基于特征变量的回归方法,当构建分类模型区分基于同一训练集的不同样品时,训练集中的每份样品会被分配1个虚拟变量作为期望值,预测集的样品分类取决于模型中的预测值Yi[18-19]。本试验是对4种转基因大豆的判别,人为的把模型响应变量期望值分别设定为:“A1”为“-1.5”;“B1”为“-0.5”;“M1”为“0.5”;“J1”为“1.5”。模型的判别阈值误差设置为±0.5,当模型对4种转基因大豆预测时,由预测值Yi大小按照Yi<-1为“A1”;-1≤Yi<0为“B1”;0≤Yi≤1为“M1”;Yi>1为“J1”的区间划分进行归类。
采用留一交互验证法来确定模型的最佳主成分数[20-21]。以交互验证均方根误差(root mean square error of cross valdarion,RMSECV)作为评价标准。表达式如公式(1)所示:
(1)
式中:n表示交互验证集样本数;ci表示第i个样本的预测值;yi表示第i个样本的期望值。
1.3.5 误差反向传播人工神经网络(back-propagation artificial neural network,BP-ANN)
ANN是通过人工建立的具有自适应、自组织、自学习特点的以有向图组成拓扑结构的动态系统。其通过正向和反向的学习和校正,实现输出和输入之间的高度的非线性映射。BP-ANN一般包含3个结构,输入层、隐含层和输出层。其包含2个过程:信号的正向传播和误差的反向传播[22-24]。
本试验为判别4种转基因大豆,由表1可知,4种转基因大豆NIR光谱数据经PCA后,前7个主成分包含了原始光谱的绝大多数信息,累计贡献率达到99.1%,所以可由前面7个主成分作为BP-ANN的输入。4种转基因大豆期望值输出可以设为:“A1”为“-3”,“B1”为“-1”,“M1”为“1”,“J1”为“3”,模型的判别阈值误差设置为±1。经过对BP-ANN的多次训练,建立了1个输入层(输入节点为7),2个隐含层(隐含层节点数分别为5和8)和1个输出层(输出节点为1)的ANN。当模型对4种转基因大豆预测时,由预测值Yi大小按照Yi<-2为“A1”;-2≤Yi<0为“B1”;0≤Yi≤2为“M1”;Yi>2为“J1”的区间划分进行归类。
1.3.6 数据处理
数据采用MATLAB软件自编的PCA、PLS-DA和BP-ANN建模程序处理,Origin 2018软件绘制图像。
2 结果与分析
2.1 转基因大豆原始光谱图
图2是240份转基因大豆样品NIR原始光谱图,由图2可知光谱带有部分噪音肉眼无法通过光谱图来区分4种转基因大豆,图3是光谱经过平滑+SNV预处理之后的光谱,由图3可知光谱的噪音明显减少,但仍然无法通过肉眼区分。
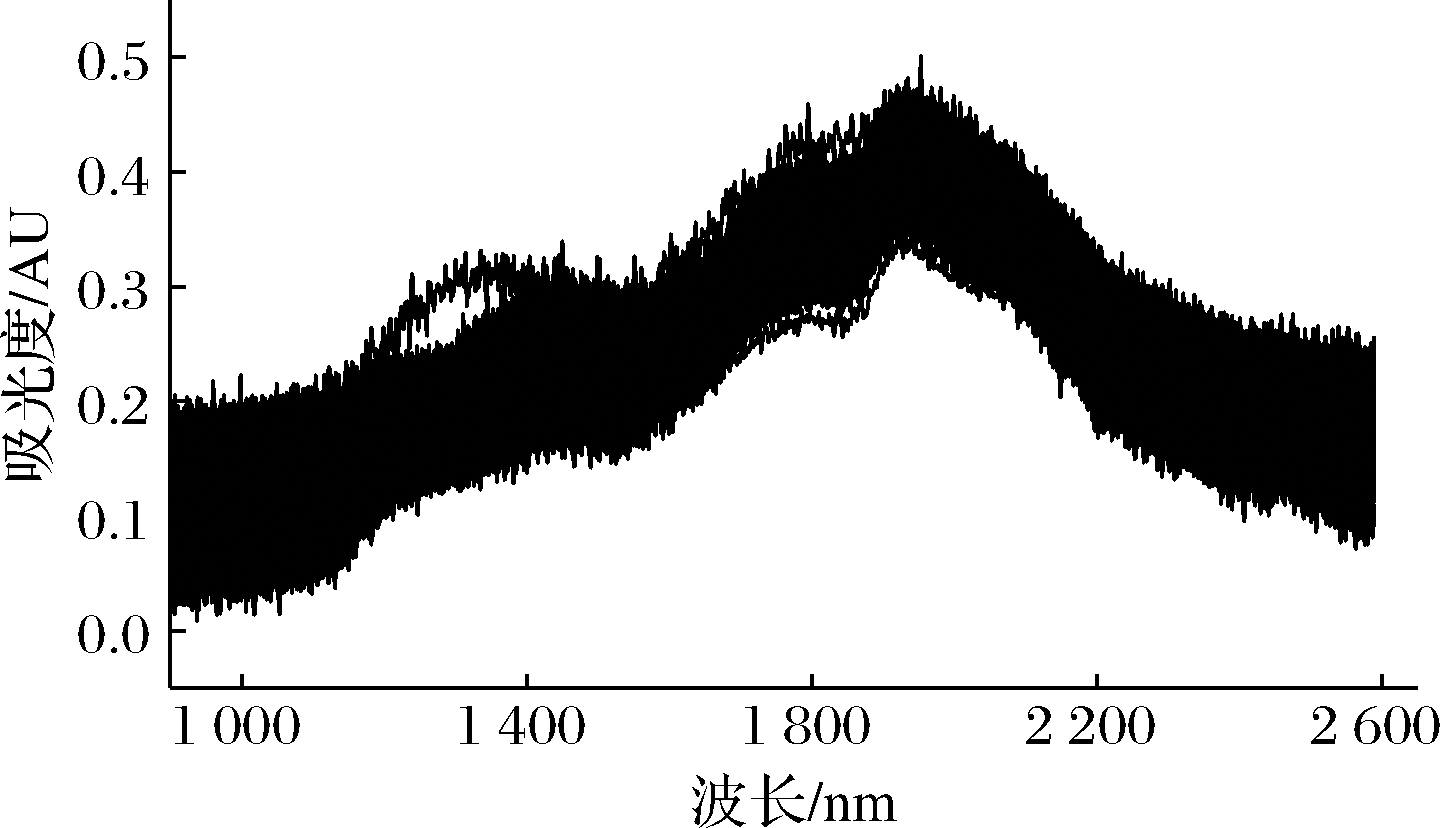
图2 转基因大豆NIR原始光谱
Fig.2 NIR spectrum of transgenic soybean
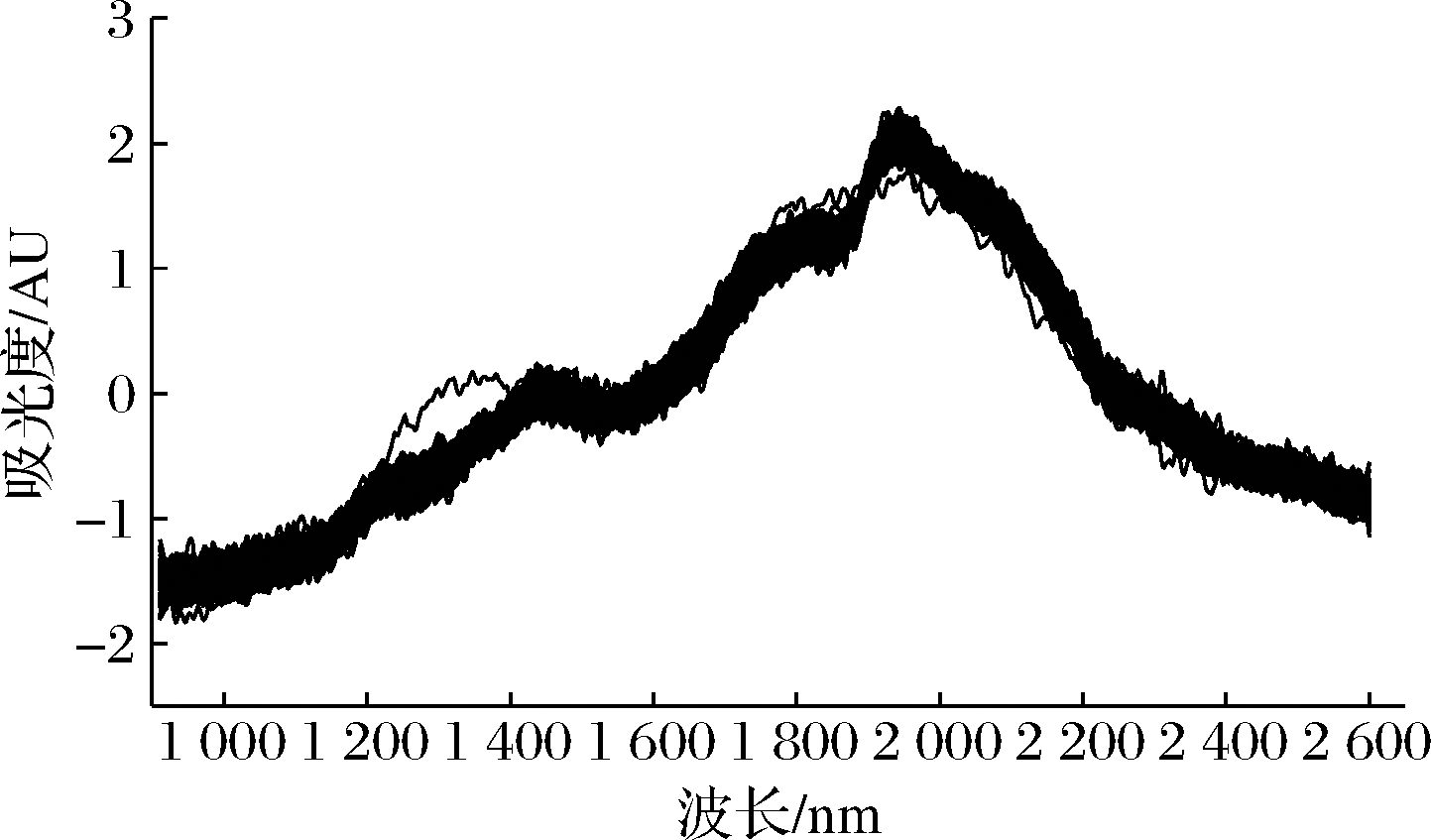
图3 平滑+SNV处理后的转基因大豆NIR光谱
Fig.3 NIR spectra of transgenic soybean after smoothing +SNV treatment
2.2 PCA判别
转基因大豆光谱经过预处理仍然有很庞大的数据。过多的冗余信息,不仅计算量大,而且还会降低模型的精度。利用PCA方法可对数据进行降维,得到各主成分的得分矩阵。
表1为NIR光谱数据中前7个主成分累计贡献率,PC1的贡献率为92.5%,PC2的贡献率为3.6%。图4为PC1和PC2的得分图,由图4可知,4种转基因大豆有较好的聚类,尤其是“A1”、“B1”和“J1”。但是“A1”和“J1”分布区域比较靠近,部分样品有覆盖,不能区分这2种转基因大豆。
表1 主成分累计贡献率
Table 1 Cumulative contribution rate of principal components


图4 主成分得分
Fig.4 Principal component score
2.3 PLS-DA
采用PLS-DA方法对4种转基因大豆判别,训练集包含180份样品和预测集包含60份样品。图5为RMSECV与选择的主成分数的关系,设置最大主成分数为20,当主成分数为4时,RMSECV最小,因此选择4为模型的最佳主成分数。
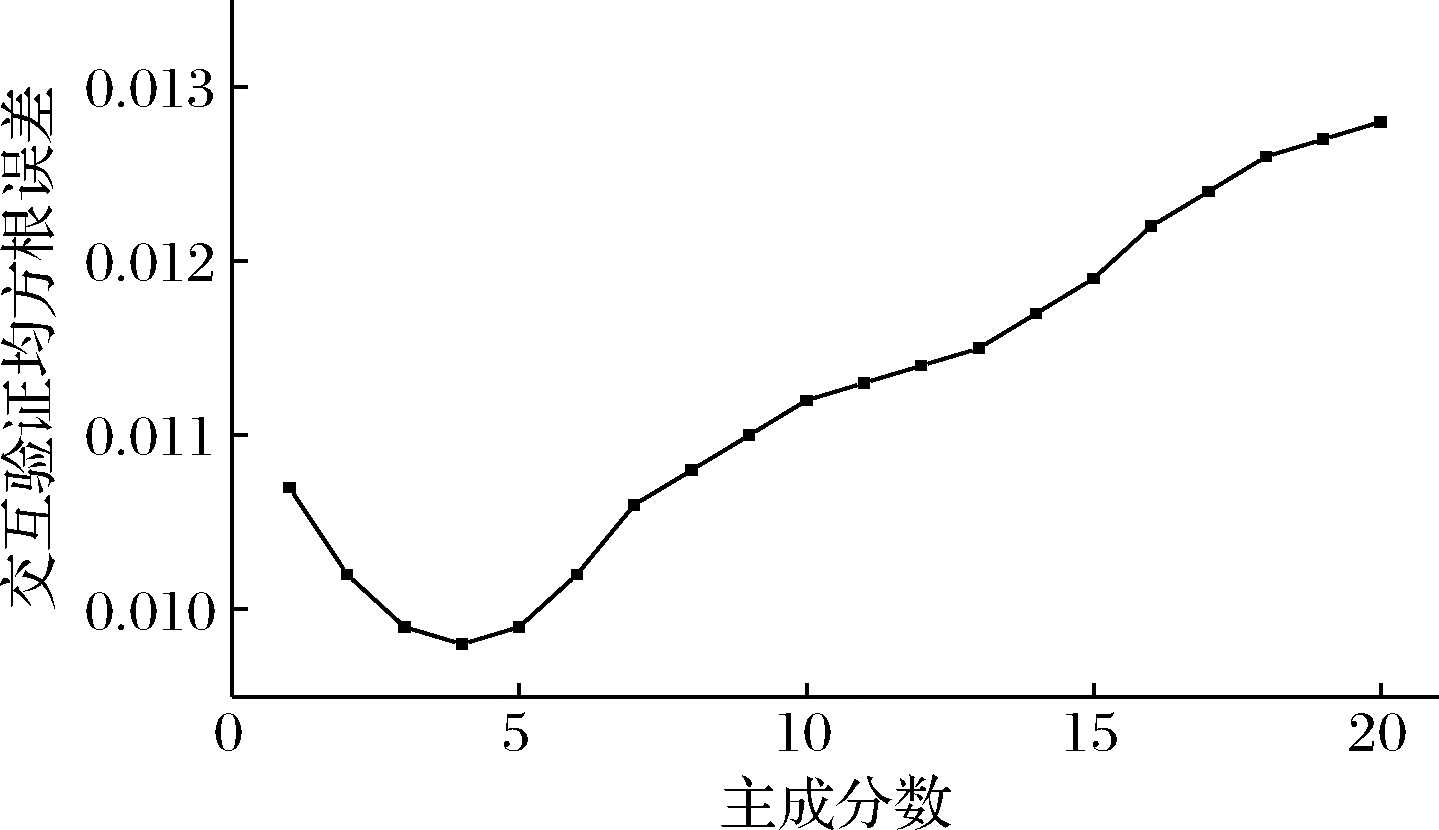
图5 RMSECV与主成分数的关系
Fig.5 Relationship between RMSECV and number of principal component
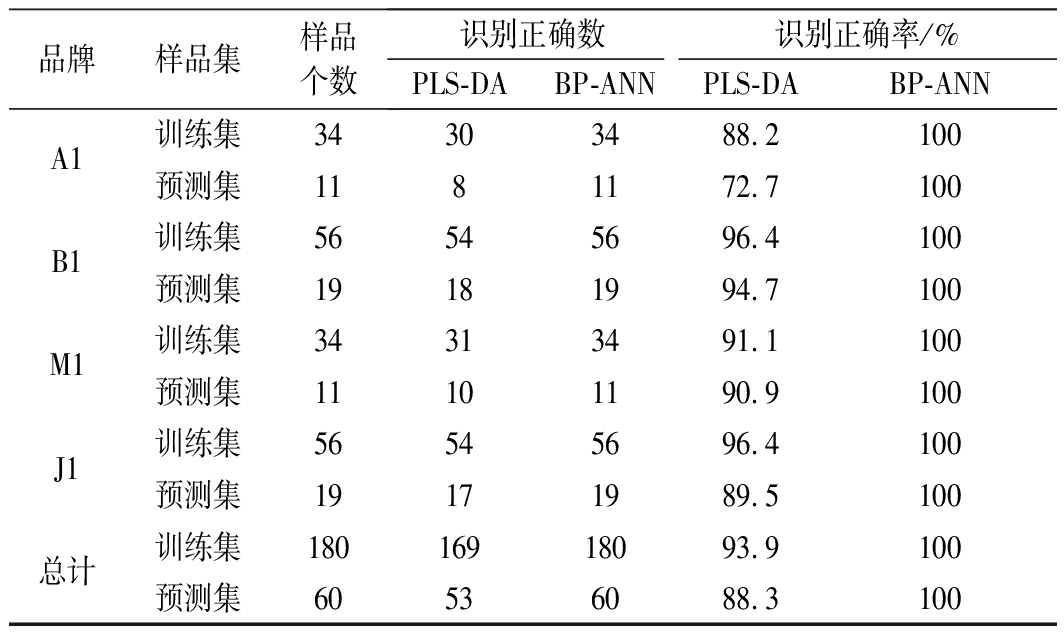
训练集样品和预测集样品的预测结果如图6和图7所示,训练集和预测集的预测结果大多数在相应的区间内。表2为4种转基因大豆的判别结果,由表2可知,训练集中“A1”的识别率为88.2%,“B1”的识别率为96.40%,“M1”的识别率为91.1%,“J1”的识别率为96.4%。预测集中“A1”的识别率为72.7%,“B1”的识别率为94.7%,“M1”的识别率为90.9%,“J1”的识别率为89.5%。4种转基因进口大豆的总识别率为92.5%。使用PLS-DA方法可以较好的识别这“B1”、“M1”和“J1”这3种转基因大豆,“A1”的识别率偏低,不能满足现实要求,需要找到更适合的方法提高“A1”的识别率。

图6 训练集转基因大豆PLS-DA方法的预测结果
Fig.6 Prediction results of PLS-DA method for transgenic soybean in training set
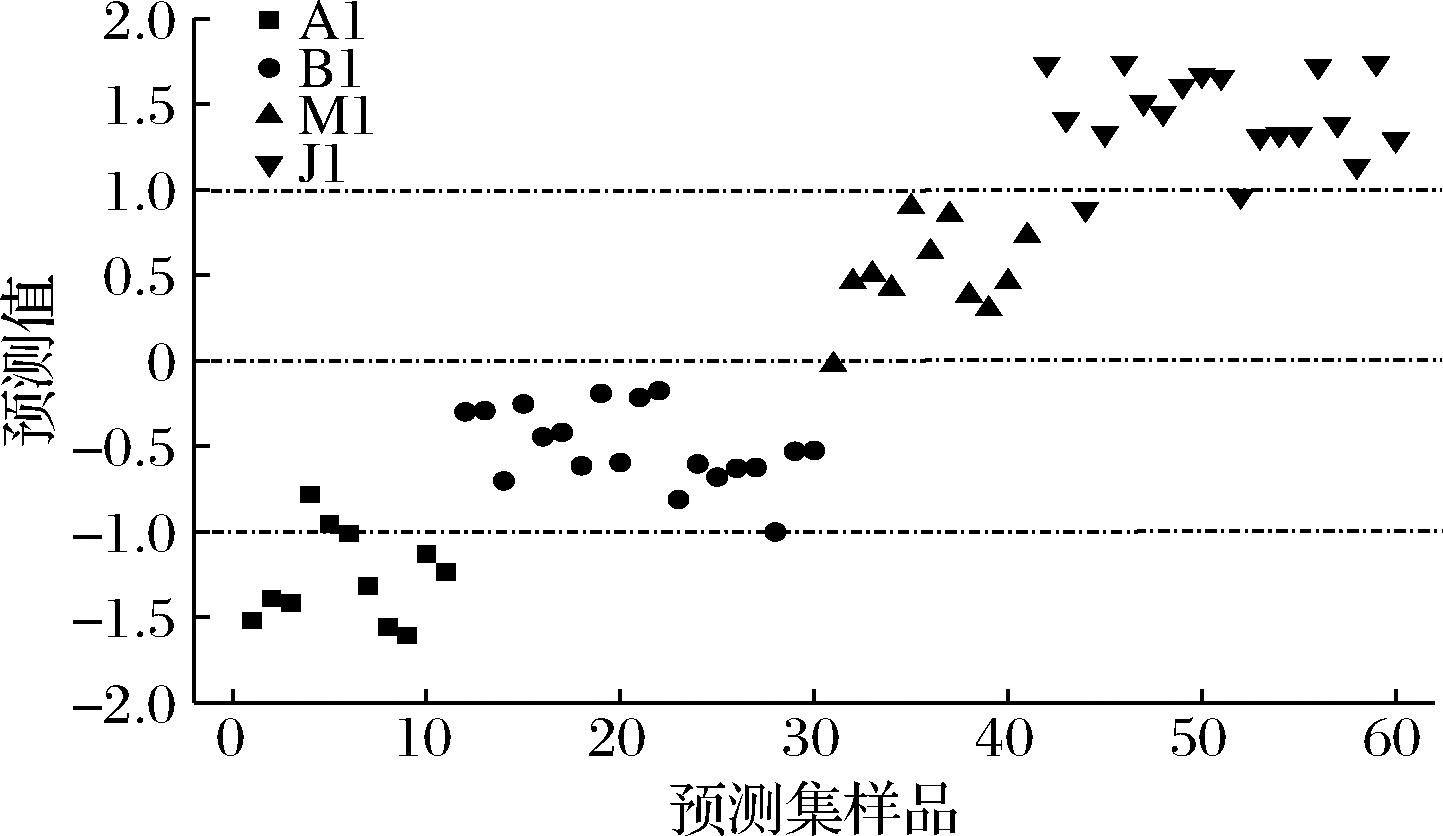
图7 预测集转基因大豆PLS-DA方法的预测结果
Fig.7 Prediction results of PLS-DA method for transgenic soybean in prediction set
表2 PLS-DA和BP-ANN方法判别结果
Table 2 Identification results for PLS-DA and BP-ANN
2.4 ANN方法判别
采用BP-ANN方法对4种转基因大豆判别,训练集包含180份样品和预测集包含60份样品,ANN经训练集训练优化后确定权值和阈值,预测集的60份样品进行验证。训练集和预测集的预测结果如图8和图9所示,训练集和预测集中“A1”、“B1”、“M1”和“J1”的预测值与期望值高度一致。由表2可知,训练集和预测集识别率均为100%。
2.5 PLS-DA模型与ANN方法判别模型的验证
取未参与建模的验证集20份样品对PLS-DA方法模型与BP-ANN方法判别模型进行验证。验证结果如图10、图11所示,PLS-DA方法模型识别率为90.0%,BP-ANN方法判别模型的识别率为100%。可见,PLS-DA方法模型与BP-ANN方法判别模型对转基因大豆识别率较高。
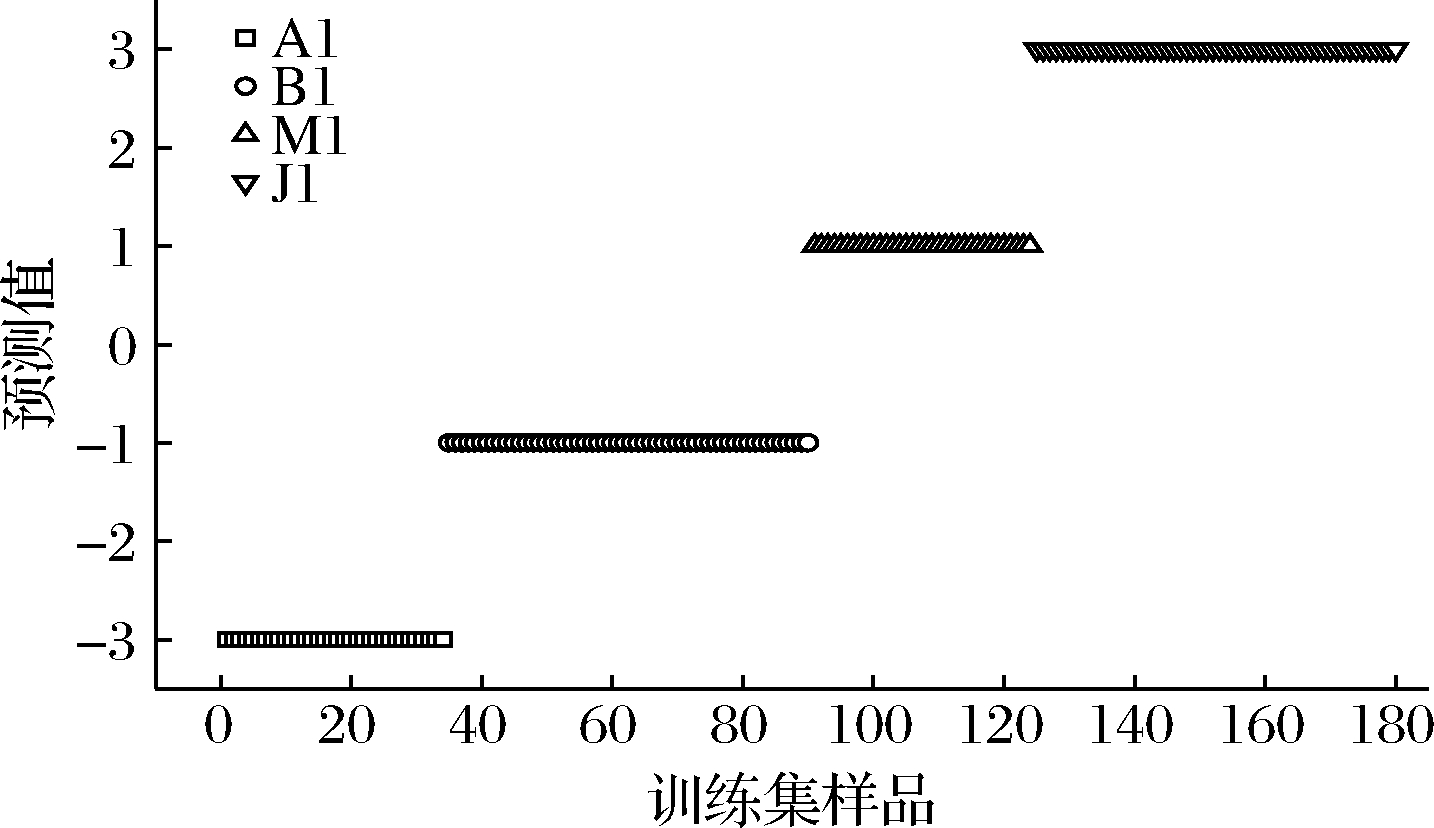
图8 训练集转基因大豆BP-ANN方法的预测结果
Fig.8 Prediction results of BP-ANN method for transgenic soybean in training set
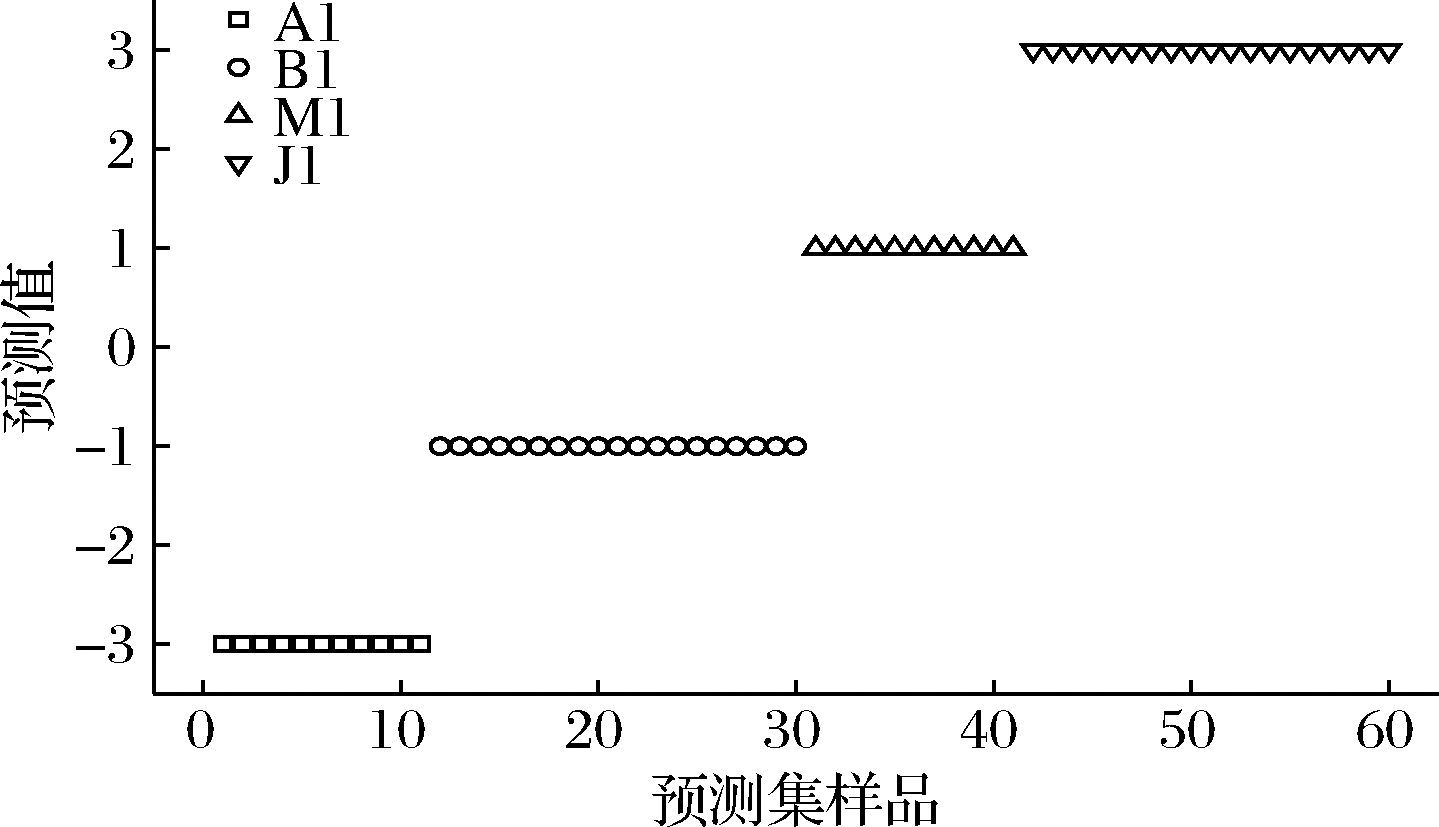
图9 预测集转基因大豆BP-ANN方法的预测结果
Fig.9 Prediction results of BP-ANN method for transgenic soybean in prediction set
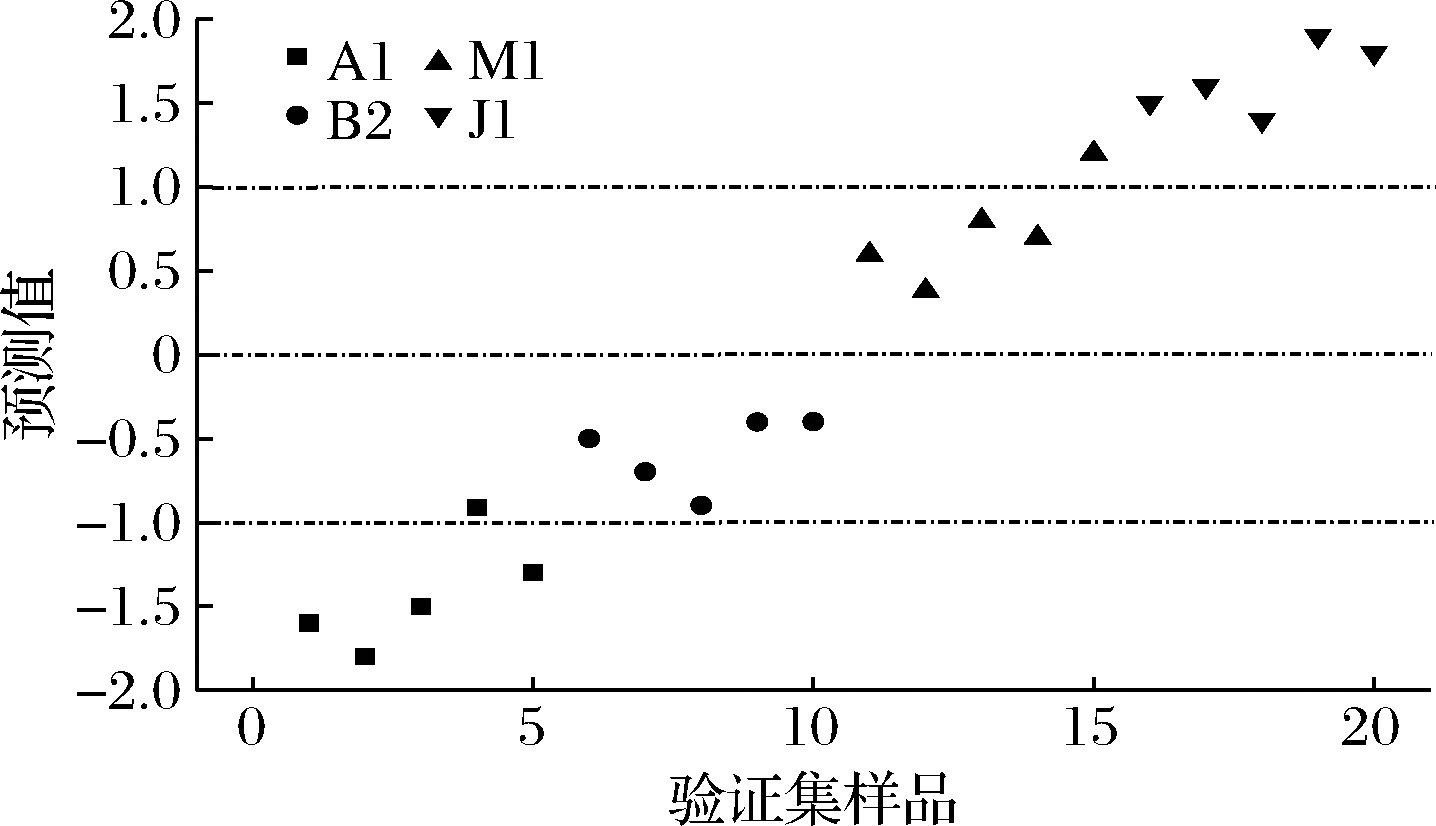
图10 验证集转基因大豆PLS-DA方法的预测结果
Fig.10 Prediction results of PLS-DA method for transgenic soybean in validation set
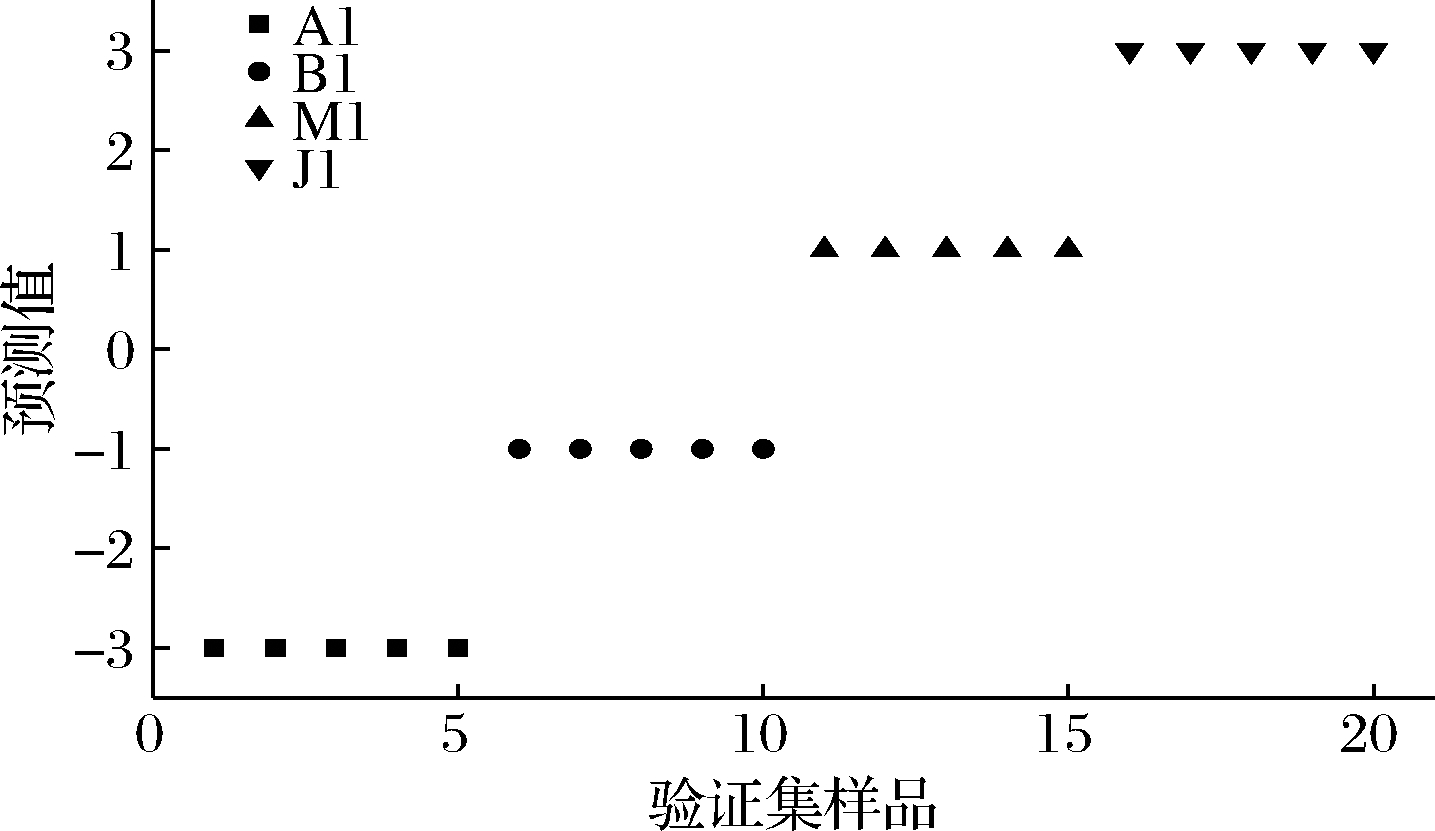
图11 验证集转基因大豆BP-ANN方法的预测结果
Fig.11 Prediction results of BP-ANN method for transgenic soybean in validation set
3 结论
采用NIR光谱结合化学计量学对4种转基因大豆进行判别分析,利用平滑+SNV方法预处理原始光谱数据,KS算法划分训练集样品和预测集样品,PCA、PLS-DA和BP-ANN方法分析预处理后的NIR光谱数据。试验结果显示平滑+SNV的预处理方法能有效减少NIR光谱的噪音;PCA方法能判别出4种转基因大豆中的3种,阿根廷转基因大豆和加拿大转基因大豆不能同时判别;PLS-DA方法对预测集转基因大豆的判别正确率为88.3%;BP-ANN方法能够准确的判别4种转基因大豆,判别正确率为100%;并用未参与建模的4种转基因大豆对PLS-DA方法模型和BP-ANN方法模型进行验证,验证集中PLS-DA方法模型判别正确率为90.0%,BP-ANN方法模型判别正确率为100%。本试验虽然只选取了4种转基因大豆进行建模判别,但是试验结果表明采用NIR光谱结合PLS-DA和BP-ANN方法对转基因大豆产地朔源是可行的。可为我国相关部门对转基因大豆的产地朔源提供部分方法。
- GB/T 7714-2025与GB/T 7714-2015相比,变更了哪些,对期刊参考文献格式有什么影响?
- 别被这个老掉牙的报纸理论版投稿邮箱误导了!最新核实91个报纸理论版投稿邮箱通道,一次集齐
- 喜报!《中国博物馆》入选CSSCI扩展版来源期刊(最新CSSCI南大核心期刊目录2025-2026版)!新入选!
- 2025年中科院分区表已公布!Scientific Reports降至三区
- 国内核心期刊分级情况概览及说明!本篇适用人群:需要发南核、北核、CSCD、科核、AMI、SCD、RCCSE期刊的学者
- CSSCI官方早就公布了最新南核目录,有心的人已经拿到并且投入使用!附南核目录新增期刊!
- 北大核心期刊目录换届,我们应该熟知的10个知识点。
- 注意,最新期刊论文格式标准已发布,论文写作规则发生重大变化!文字版GB/T 7713.2—2022 学术论文编写规则
- 盘点那些评职称超管用的资源,1,3和5已经“绝种”了
- 职称话题| 为什么党校更认可省市级党报?是否有什么说据?还有哪些机构认可党报?

 0373-5939925
0373-5939925 2851259250@qq.com
2851259250@qq.com



